京都大学と東京大学の共同研究グループは28日、遺伝子をノックアウトした後の時系列発現の変化を計測したデータから、数理モデルに基づいて遺伝子制御ネットワークを高精度かつ半自動的に推定する手法「RENGE」を開発したと発表した。同日付で国際学術誌「コミュニケーションズバイオロジー」に掲載されている。
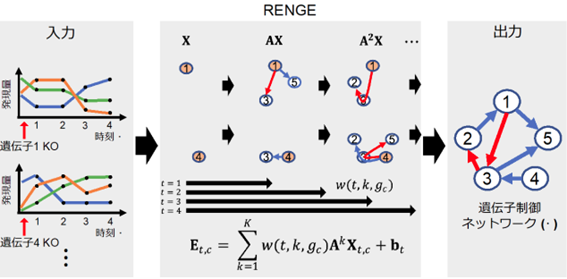
1細胞CRISPR実験により得られるデータからノックアウト前後での発現変化を解析するだけでは、その遺伝子から直接制御されているものと、間接的に制御されているものを区別することはできない。ノックアウトすればその下流にある遺伝子も変化するからだ。
研究チームは、ノックアウトした後に時系列で発現量を計測し、その発現ダイナミクスから情報を抽出することで、問題を解決できる可能性に気づき、情報から遺伝子制御ネットワークを推定するRENGEを開発した。
仮想的なデータを用いて、RENGEの推定精度を検証したところ、既存手法より際立って優れた性能を示した。次に、ヒトiPS細胞を用いて1細胞CRISPR実験を行い、遺伝子ノックアウト後の時系列発現データを取得。
このデータにRENGEを適用することで、人の多能性維持に関わる103遺伝子を含む遺伝子制御ネットワークを発見。また、これを解析した結果、これまでに知られていた多能性維持に働く遺伝子制御ネットワークの特徴が再現されていた。
研究グループの石川雅人京都大学特定助教は「新しい実験技術とそれに適した数理的解析手法を組み合わせることで、生命システムを効率的に解明していくことができる」と評価している。
基づき、遺伝子制御ネットワークを推定
